La psoriasis vulgar y la artritis psoriásica son trastornos relacionados entre sí, con un importante componente genético. Aunque los estudios de ligamiento han llevado a la identificación de diversos loci y genes de susceptibilidad, ha sido el reciente progreso tecnológico y la realización de estudios de asociación genómica extensos lo que ha permitido demostrar asociaciones robustas de la psoriasis con diversos genes, asociados o no al complejo mayor de histocompatibilidad. La mayoría de estos genes se pueden incorporar en un modelo patogénico integrado que comprende distintas redes de señalización que afectan la función barrera de la piel (LCE3, DEFB4, GJB2), la respuesta inmune innata implicando al sistema de señales del factor nuclear-κB (TNFAIP3, TNIP1, NFKBIA, REL, FBXL19, TYK2, NOS2, CARD14), y la respuesta inmune adaptativa implicando a linfocitos T CD8 y las señales de la vía interleucina 23 (IL-23)/IL-17 (HLA-C, IL12B, IL23R, IL23A, TRAF3IP2, ERAP1). La mejor comprensión de las potenciales interacciones entre los genes implicados y de estos con factores ambientales, así como el conocimiento de las alteraciones en las funciones de las proteínas codificadas tendrán sin duda implicaciones nosológicas, terapéuticas y pronósticas.
Psoriasis vulgaris and psoriatic arthritis are interrelated disorders with an important genetic component. While linkage studies have identified several candidate loci and genes, only recent technological advances and extensive genome-wide association studies have provided robust evidence of associations between psoriasis and several genes inside and outside the major histocompatibility complex. Most of these genes can be incorporated into an integrated pathogenic model of psoriatic disease comprising distinct signaling networks affecting skin barrier function (LCE3, DEFB4, GJB2), innate immune responses involving nuclear factor-κB signaling (TNFAIP3, TNIP1, NFKBIA, REL, FBXL19, TYK2, NOS2, CARD14), and adaptive immune responses involving CD8 T cells and interleukin 23 (IL-23)/IL-17-mediated lymphocyte signaling (HLA-C, IL12B, IL23R, IL23A, TRAF3IP2, ERAP1). A better understanding of the potential gene/gene and gene/environment interactions and of the functions of altered transcripts will undoubtedly have nosologic, therapeutic and prognostic implications.
La psoriasis vulgar no es una enfermedad genéticamente homogénea y existen evidencias de que los diferentes subfenotipos de la enfermedad pueden estar asociados a diferentes variaciones genéticas. Por ejemplo, la pustulosis palmo-plantar pura se considera una entidad independiente de la psoriasis vulgar, que a su vez tendría una asociación genética más próxima a la psoriasis guttata1. La psoriasis vulgar es una enfermedad inflamatoria crónica que presenta una clara asociación con determinados alelos del gen HLA-C y, en concreto, con el alelo HLA-Cw6 (esto es, HLA-Cw*0602 cuando se identifica mediante genotipado de alta resolución), presente en un 30% de los pacientes (en comparación con el 10-15% de la población general). El riesgo relativo de presentar la enfermedad en los pacientes homozigotos es 2,5 veces superior al de los pacientes heterozigotos2. Los pacientes HLA-Cw6 positivos tienen unas determinadas características clínicas definidas por un inicio precoz de la enfermedad, la presencia de placas más extensas y una mayor incidencia de fenómeno de Koebner. Asimismo también se ha descrito una mayor frecuencia de faringoamigdalitis estreptocócica como factor desencadenante, una mayor respuesta a la luz solar y un curso en ocasiones más grave. En los pacientes HLA-Cw6 negativos se ha descrito una mayor frecuencia de alteraciones ungueales y de artritis psoriásica2,3.
Antes de exponer en detalle los aspectos genéticos mas importantes de la enfermedad revisaremos sus bases inmunopatogénicas, para poder dar finalmente una visión más integradora que nos proporcione una base teórica de los grandes avances terapéuticos que se han producido en los últimos años, y que serán objeto de discusión detallada en los siguientes apartados.
Inmunopatogenia de la psoriasisLa psoriasis se caracteriza por una proliferación epidérmica marcada y un trastorno de la diferenciación con activación inmune de los queratinocitos, que se acompaña de numerosas alteraciones de naturaleza inflamatoria e inmunológica, con participación tanto de la inmunidad innata como de la adquirida4–6. Intervienen de forma fundamental los linfocitos T, como sugirieron hace ya 25 años los datos de eficacia de la ciclosporina7 y confirmaron posteriormente los resultados terapéuticos de inmunomoduladores selectivos de los linfocitos T8–10. También se ha demostrado recientemente la marcada eficacia de los agentes biológicos dirigidos contra el factor de necrosis tumoral alfa (TNF-α)11, que interviene como mediador pleotrópico de la inflamación en diversos órganos, así como de los anticuerpos anti-p40, que bloquean la diferenciación y expansión de los linfocitos Th1 y Th17 a través de la interleucina 12 (IL-12) y la IL-23, respectivamente12.
El papel del TNF-α y de los linfocitos T residentes en la piel se ha comprobado en un modelo experimental con ratones AGR129, que carecen de receptores para interferón (IFN) y de células natural killer (NK), por lo que son incapaces de rechazar la piel humana13. Cuando se injerta piel aparentemente indemne de pacientes con psoriasis se desarrollan espontáneamente placas de psoriasis, sin necesidad de añadir linfocitos T CD4+. El examen de biopsias seriadas demuestra que los linfocitos T humanos residentes en los injertos proliferan y producen TNF-α, y el tratamiento con anticuerpos anti-CD3 humano, que impide la proliferación de los linfocitos T, o inhibidores del TNF-α (infliximab o etanercept) previene la conversión de las placas pre-psoriásicas en lesiones de psoriasis13. Por otra parte, cuando se bloquea la exocitosis de los linfocitos T a la epidermis mediante un anticuerpo contra la integrina α1β1 se inhibe este proceso. Cuando ya existen linfocitos T en la epidermis la inhibición es parcial, y el tratamiento resulta inefectivo cuando se injertan lesiones completamente desarrolladas de psoriasis, lo que confirma el papel de los linfocitos T residentes y su emigración a la epidermis en el desarrollo de las lesiones de psoriasis14.
El reciente descubrimiento de una subpoblación de linfocitos T que expresan IL-17, y cuya expansión viene determinada por la acción de la IL-23 producida por las células presentadoras de antígeno y las células dendríticas sobre los precursores T naïve15, ha revolucionado la patogenia de la psoriasis. Se ha demostrado una marcada expansión de los linfocitos T citotóxicos que expresan de forma independiente IL-17 e IL-22 en la epidermis psoriásica16,17. La expansión de los linfocitos Th1 retroalimentaría este proceso, al estimular la síntesis de IL-12 e IL-23 por las células presentadoras de antígeno mediante la producción de IFN γ16.
Estudios de ligamiento genético de la psoriasisEn los años 90 diversos grupos iniciaron estudios de ligamento genético analizando la cosegregación de marcadores genéticos tipo microsatélite en familias con diferentes individuos con psoriasis. Mediante estos estudios se han mapeado por lo menos 6 loci de susceptibilidad a la psoriasis, denominados PSORS1 a PSORS618–21. El principal determinante genético de la psoriasis (PSORS1), localizado en la región cromosómica 6p21, que contribuye a entre un 30 y un 50% de la susceptibilidad genética a la enfermedad, probablemente corresponda al alelo HLA-Cw*0602, aunque esto no explicaría los casos de psoriasis de inicio tardío20. Se ha postulado que este alelo permitiría la presentación de un epítopo putativo presente en las queratinas de tipo i, en concreto en aquellas cuya expresión está aumentada en la psoriasis. Este epítopo actuaría como un autoantígeno, presentaría reactividad cruzada con la proteína M estreptocócica y perpetuaría una respuesta autoinmune mediada por los linfocitos CD8+ (y tal vez por linfocitos CD4+ con receptores NK-T), capaces de reconocer al complejo mayor de histocompatibilidad de tipo i, y dando lugar a la cronificación de las lesiones19. El segundo locus de susceptibilidad a la psoriasis, PSORS2, identificado mediante análisis de ligamiento en diferentes familias, se localiza en la región 17q24-q25, donde también se ha identificado un locus de susceptibilidad para la dermatitis atópica, y los genes putativos estarían implicados en la regulación de la sinapsis inmunológica3,21. En estudios de ligamiento realizados en familias de diversa procedencia geográfica se han identificado otros loci (tabla 1) tales como PSORS 3 (4q34), PSORS 4 (1q21), PSORS 5 (3q21), PSORS 6 (19p13), PSORS 7 (1p32), PSORS 9 (4q31), y se están investigando otros21,22, aunque el ligamiento con PSORS1 es claramente el más importante. Recientemente se ha confirmado la identificación del alelo HLA-Cw6 como el locus responsable de la asociación en PSORS123, y este hallazgo se ha confirmado en un estudio extenso de una población étnicamente diferente24.
Loci asociados a la psoriasis (PSORS) y artritis psoriásica (PSORSA)
| Locus | Región | OMIM | Genes candidatos/función |
| PSORS1 | 6p21.3 | 612410 | HLA-Cw6 |
| PSORS2 | 17q25.5-qter | 607211 | CARD14 |
| PSORS3 | 4q34 | 601454 | IRF-2 |
| PSORS4 | 1q21 | 603935 | Loricrina, filagrina, Pglyrp3,4; genes S100 y late cornified envelope (en el complejo de diferenciación epidérmica) |
| PSORS5 | 3q21 | 604316 | SLC12A8, cistatina A, proteína con dedo de cinc 148 |
| PSORS6 | 19p13 | 605364 | JunB |
| PSORS7 | 1p | 605606 | PTPN22 (1p13), IL23R (1p32.1-31.2) |
| PSORS8/PSORSA1 | 16q | 610707 | CX3CL1, CX3R1, NOD2/CARD15 |
| PSORS9 | 4q31 | 607857 | IL15 |
| PSORS10 | 18p11 | 612410 | |
| PSORS11 | 5q31-q33 | 612599 | IL12B |
| PSORS12 | 20q13 | 612950 | ZNF313/RNF114, ligasa de ubicuitina |
| PSORS13 | 6q21 | 614070 | TRAF3IP2 |
Fuente: modificada de Duffin et al.48.
El reciente desarrollo de la investigación genética basada en el conocimiento de millones de polimorfismos de un solo nucleótido (Single Nucleotide Polymorphisms [SNP]) utilizados como marcadores genéticos, el mapeado sistemático de haplotipos humanos y el desarrollo de plataformas de genotipado de alto rendimiento ha permitido efectuar estudios de asociación genómica (Genomewide Association Studies [GWAS]). En los estudios GWAS se genotipan miles o incluso millones de marcadores SNP por cada individuo, de forma que se obtiene>90% de la variación común presente en el genoma humano. Puesto que se analizan un gran número de marcadores, y en muchos casos los efectos genéticos son moderados (odds ratio [OR]<2) este tipo de estudios genéticos requieren grandes cohortes de pacientes y controles. Por lo que respecta a la psoriasis, los principales loci definidos por un efecto genético con un OR >1,25 son HLA-C, IL12B, IL23R, IL23A, IL4/IL13, TNFAIP3, TNIP1, TRAF3IP2, TYK2 e IFIH, aunque otros loci están en fase de identificación y validación (tabla 2). Algunos de estos loci asociados a la psoriasis se ha visto que también confieren susceptibilidad a otras enfermedades inflamatorias de naturaleza inmune, y son sugestivos en otros casos de variaciones étnicas en la enfermedad25,26.
Genes asociados con la psoriasis no incluidos en el MHC (generalmente identificados mediante estudios de GWAS empleando SNP o variaciones del número de copias). Las correspondientes referencias aparecen en el texto
| Gen candidato | Región/superposición con PSORS | OMIM | Función propuesta | Pleotropismo (diferentes enfermedades con las que se ha asociado) |
| IL23R | 1p31.3 (PSORS7) | 607562 | Codifica el receptor de IL-23 | Psoriasis, Crohn, colitis ulcerosa, espondilitis anquilosante |
| IL12B | 5q33.3 | 161561 | Codifica la subunidad p40 de IL-12 e IL-23 | Psoriasis, artritis psoriásica |
| IL13 | 5q31.1 | 147683 | Codifica IL-13; próximo a IL4, IL5 y el complejo RAD50 | Psoriasis, artritis psoriásica, asma, atopia |
| IL23A | 12q13.3 | 605580 | Codifica la subunidad p19 de IL-23 | Psoriasis, artritis psoriásica |
| TNFAIP3 | 6q23.3 | 191163 | Codifica la proteína A20, que actúa a través de ubicuitina inhibiendo la activación proinflamatoria de NFκB inducida por TNF-α | Psoriasis, artritis psoriásica, Crohn, artritis reumatoide lupus eritematoso sistémico, diabetes tipo1, celiaquía |
| TNIP1 | 5q33.1 | 607714 | Codifica la proteína ABIN-1, que reduce la activación proinflamatoria de NFκB inducida por TNF-α | Psoriasis, artritis psoriásica |
| TRAF3IP2 | 6q21 | 607043 | Codifica una proteína que interviene en la señalización de IL-17 e interacciona con miembros de la familia de factores de transcripción Rel/NFκB | Psoriasis, artritis psoriásica |
| ZNF313/RNF114 | 20q13 (PSORS12) | 612451 | Codifica una ligasa de ubicuitina que se expresa en la piel | Psoriasis |
| ADAM33 | 20p13 | 607114 | Disintegrina y metaloproteasa 33 | Psoriasis, asma |
| PTPN22 | 1p13.2 (PSORS7) | 600716 | Tirosín fosfatasa que interviene en la señalización de los receptores de linfocitos T | Psoriasis, artritis reumatoide, lupus eritematoso sistémico |
| CDKAL1 | 6p22 | 611259 | Codifica una proteína homóloga a una proteína cinasa | Psoriasis, Crohn, diabetes tipo 2 |
| KIR2DS1, KIR2DL1 | 19q13.4 | 604952, 604936 | Codifican receptores semejantes a inmunoglobulina que se unen a HLA-C y regulan la respuesta de las células NK | Psoriasis, artritis psoriásica |
| LCE3D/LCE3A LCE3C_LCE3B_del | 1q21 (PSORS4) | 612616, 612613 | Codifican proteínas del envoltorio de cornificación tardía (LCE), altamente expresadas en la psoriasis | Psoriasis |
| DEFB4 | 8p23.1 | 602215 | Codifica la β-defensina humana | Psoriasis |
| IL15 | 4q31.2-q32.1 (PSORS9) | Codifica una interleucina que afecta la activación y proliferación de los linfocitos T | Psoriasis | |
| IL2, IL21 | 4q27 | 147680, 605384 | Codifican interleucinas que intervienen en la proliferación de los linfocitos T, la diferenciación de los Th17 y la proliferación queratinocitaria | Artritis psoriásica, artritis reumatoide, diabetes tipo1, colitis ulcerosa |
| IL28RA | 1p36.11 | 607404 | Codifica subunidad alfa del receptor de IL-28 | Psoriasis |
| REL | 2p16.1 | 164910 | Codifica un oncogén miembro de la familia de factores de transcripción Rel/NFκB | Psoriasis |
| IFIH1 | 2q24.2 | 606951 | Codifica una helicasa inducida por interferón | Psoriasis |
| ERAP1 | 5q15 | 606832 | Codifica una aminopeptidasa del retículo endoplásmico; interviene en el procesamiento de péptidos por MHC-I | Psoriasis en pacientes positivos para HLA-Cw*0602; inicio precoz en chinos de la etnia han |
| NFKBIA | 14q13.2 | 604495 | Codifica una proteína que inactiva NFκB secuestrándolo en el citoplasma | Psoriasis |
| TYK2 | 19p13.2 | 176941 | Codifica una proteína que interviene en la señalización del receptor de interferón de tipo i | Psoriasis, lupus eritematoso sistémico |
| PTTG1 | 5q33.3 | 604147 | Interviene en proliferación y transformación celular | Psoriasis |
| CSMD1 | 8p23.2 | 608397 | Producto interviene en la activación del complemento (?) | Psoriasis |
| GJB2 | 13q12.11 | 121011 | Conexina 26 (queratodermias) | Psoriasis |
| SERPINB8 | 18q22.1 | 601697 | Inibidor de la proteasa 8 (regula múltiples funciones); aumento de expresión en lesiones de psoriasis | Psoriasis |
| ZNF816A | 19q13.41 | Codifica una proteína con dedo de cinc (intervienen en el reconocimiento de ARN y otras proteínas) de la misma clase que el producto de ZNF313 | Psoriasis de inicio precoz en chinos de la etnia han | |
| NOS2 | 17q11.2 | 163730 | Sintetasa de óxido nítrico | Psoriasis, artritis psoriásica, hipertensión, malaria |
| FBXL19 | 16p11.2 | 609085 | Ligasa de ubicuitina | Psoriasis, artritis psoriásica |
| PSMA6 (?) | 14q13.2 | 602855 | Subunidad del proteasoma (regula inflamación a través de NFκB) | Psoriasis, artritis psoriásica, susceptibilidad a infarto de miocardio |
| CARD14 | 17q25.3-qter | 607211 | Activación de NFκB; interviene en la apoptosis |
GWAS: estudios de asociación genómica; MHC: complejo mayor de histocompatibilidad; SNP: polimorfismos de un solo nucleótido.
Los GWAS han proporcionado evidencia genética de la implicación de la vía de la IL-23 en la psoriasis. El primer estudio de asociación genética a gran escala en la psoriasis (que no era un GWAS en sentido estricto, porque solo se analizaron SNP dentro o cerca de genes, pero no en otras regiones del genoma) permitió identificar la asociación con un SNP situado en la región 3’ terminal del gen IL12B. Este gen codifica la subunidad p40 común a las interleucinas IL-12 y IL-23, y fue el primer locus de riesgo de la psoriasis independiente del complejo mayor de histocompatibilidad (MHC) asociado de forma clara y reproducible27. Un estudio posterior ha permitido la identificación de un segundo polimorfismo asociado de forma independiente a la enfermedad. A su vez, se han identificado 2 SNP en el locus que codifica una de las subunidades del receptor de la IL-23 (IL23R), que también muestran una asociación independiente con la psoriasis28. La asociación de estos loci ha sido validada en diferentes grupos poblacionales29–31, tanto en la psoriasis como en la artritis psoriásica32,33. Asimismo se ha demostrado la asociación del gen IL23A, que codifica la subunidad p19 de la IL-23, con la psoriasis y la artritis psoriásica28, además de la espondilitis anquilosante y la enfermedad de Crohn, pero no con la artritis reumatoide o la enfermedad celiaca33.
El segundo GWAS publicado34 confirmó la asociación de los genes IL12B e IL23R con la psoriasis y también con la artritis psoriásica. Se identificó una nueva señal próxima al gen IL23RB2, así como nuevos loci candidatos en las regiones 13q13, que contienen los genes COG6 y LHFP; 15q21, que contiene el gen TNFAIP8L3; 4q27, que contiene los genes IL2 e IL21, y 1q21 con el gen LCE1C. Sin embargo, el bajo tamaño muestral empleado en este estudio no permitió asociar de forma inequívoca estos genes con la psoriasis.
En un GWAS llevado a cabo en una gran población de chinos de etnias han y uigur35, incluyendo tanto un análisis de SNP como de variaciones en el número de copias (esto es, copy number variants [CNV]), se identificó una nueva asociación con el cluster de genes del envoltorio tardío de cornificación (late cornified envelope [LCE]) en el cromosoma 1q21, previamente identificado como PSORS4 mediante estudios de ligamiento. Los productos de codificación de los genes de esta región intervienen en la diferenciación terminal de la epidermis, lo que hace de dichos genes excelentes candidatos para explicar diversos fenotipos de la psoriasis. Un estudio publicado en paralelo identificó una CNV en esta misma región cromosómica asociada también a psoriasis. Esta CNV es una deleción (es decir, ausencia de un segmento de ADN) y presenta una alta correlación (o desequilibrio de ligamiento) con el SNP rs4112788, pero no con el SNP rs6701216, publicada por Liu et al.36. Esta asociación con SNP correlacionados con la deleción LCE3C_LCE3B-del se ha confirmado en una población de pacientes con artritis psoriásica británicos36, pero no en una población de pacientes alemanes37. En este caso, la heterogeneidad fenotípica de cada cohorte y el reducido tamaño muestral pueden explicar las diferencias observadas.
Los genes no actúan de forma aislada, sino que operan a través de redes moleculares complejas y participan en diversas vías celulares. De la misma forma, la asociación de determinadas variaciones genéticas con el riesgo de desarrollar una enfermedad puede estar condicionada por la presencia de otras variaciones presentes en el genoma. La interacción génica, o epistasis, es un mecanismo genético complejo del que hasta hace muy poco no existían evidencias robustas en humanos. Resulta relevante que las principales evidencias de la existencia de epistasis en enfermedades humanas hayan sido identificadas en la psoriasis. En una población china se identificó la presencia de interacciones epistáticas entre el MHC y otros genes de riesgo para la psoriasis, como LCE e IL12B38. El riesgo de psoriasis en los individuos que tienen los alelos de riesgo en MHC y LCE aumenta 26 veces y, en el caso de MHC e IL12B, 36 veces con respecto a los individuos que no los tienen. Sin embargo, en un estudio efectuado en una población del norte de China se ha observado que la asociación con la deleción LCE3C_LCE3B-del depende de la edad de inicio y de los antecedentes familiares del paciente, y no presenta epistasis (modificación de la susceptibilidad) con el alelo HLA-Cw639.
En 2006 se estableció una colaboración entre los investigadores de la Universidad de Michigan, la Universidad Washington en Saint Louis y la Universidad de Utah para llevar a cabo estudios GWAS en psoriasis. En 2009 se publicaron los resultados del primer GWAS de este consorcio, conocido como Collaborative Association Study of Psoriasis (CASP). En este estudio se genotiparon 438.670 SNP en 1.409 casos y 1.436 controles en una primera fase, seguida del genotipado de los 21 SNP con mayor evidencia estadística de asociación, correspondientes a 18 loci, en una cohorte independiente de 5.048 casos y 5.041 controles28. En este estudio se encontró evidencia de asociación (p<0,05 en la cohorte de seguimiento) para 10 loci, que fue especialmente convincente (p<0,0005 en la cohorte de seguimiento) para 7 de ellos, confirmándose la asociación con HLA-Cw6, IL13, IL12B y IL23R, e identificándose la mencionada asociación con IL23A. Un hallazgo novedoso de este estudio fue la asociación con 2 genes de la vía del factor de señalización del factor de transcripción NF-κB (implicada en la patogénesis de enfermedades autoinmunes como el lupus eritematoso sistémico o la artritis reumatoide): TNF-α induced protein 3 (TNFAIP3) y TNFAIP3 interacting protein 1 (TNIP1). En este estudio no se encontraron diferencias en cuanto a las asociaciones en función de la presencia de artritis psoriásica, a diferencia de otros estudios en los que IL13 parece asociarse específicamente a la artritis psoriásica35,40. Recientemente se han publicado diversos GWAS en poblaciones independientes que confirman la asociación del gen TRAF3IP2 tanto con la psoriasis como con la artritis psoriásica41,42. Este gen codifica una proteína que interviene en las señales inducidas por IL-17 e interacciona con diversos miembros de la familia de factores de transcripción Rel/NF-κB.
Se han identificado asimismo de forma repetida mediante GWAS o análisis de SNP otras señales en diferentes poblaciones43–45, por ejemplo un locus próximo al gen ZNF313/RNF114, en 20q13, que —como TNFAIP3 y TNIP1— codifica una ligasa de ubicuitina46, o en las regiones de los genes CDKAL1, PTPN22 y ADAM3347, que se han confirmado en el caso del primero26, asimismo implicado en la susceptibilidad a la diabetes tipo 2 y a la enfermedad de Crohn48,49.
Uno de los últimos estudios publicados refuerza la hipótesis de que en la patogenia de la psoriasis se integran determinantes genéticos de una disfunción de la barrera epidérmica, con una alteración de la regulación de la inmunidad innata y adaptativa. En el Genetic Analysis of Psoriasis Consortium y el Wellcome Trust Case Control Consortium 250 se llevo a cabo un GWAS con 594.224 SNP en 2.622 pacientes con psoriasis y 5.667 controles, confirmándose la asociación con TRAF3IP2 e identificándose 7 nuevos loci que contienen genes con funciones inmunes: IL28RA, REL, IFIH1, ERAP1, NFKBIA y TYK2. Estas asociaciones se validaron en una cohorte de replicación de 9.079 muestras de individuos caucásicos europeos51. En este estudio se identificó la asociación epistática de los genes HLAC y ERAP1 con el riesgo de padecer psoriasis. El producto del gen ERAP1 interviene en el procesamiento de péptidos por el MHC de clase i y el alelo de riesgo de dicho gen solo aumenta el riesgo de psoriasis en aquellos individuos que son positivos para el alelo HLA-Cw*060251, siendo uno de los primeros ejemplos claros y reproducibles de epistasis en humanos.
Un reciente estudio de replicación52 del GWAS, realizado en población china37, incluyendo 8.312 pacientes con psoriasis y 12.919 controles chinos, 3.293 casos y 4.188 controles de Alemania y Estados Unidos y 254 familias nucleares de Estados Unidos, ha permitido identificar 6 nuevos loci de susceptibilidad que contienen los genes candidatos ERAP1, PTTG1, CSMD1, GJB2, SERPINB8 y ZNF816A y han replicado un locus en 5q33.1 (TNIP1-ANXA6), previamente publicado en estudios europeos. Dos de los loci identificados (ZNF816A y GJB2) también presentan evidencia de asociación en el estudio en población Alemana. Por otra parte, ERAP1 y ZNF816A se asocian con la psoriasis de tipo 1 (es decir,. psoriasis de inicio precoz) en la población china de etnia han. Aparte de identificar nuevos factores de susceptibilidad genética, este estudio ilustra claramente que parte la heterogeneidad genética presente en la enfermedad se debe a diferencias genéticas entre poblaciones étnicamente diferentes.
Los resultados de los estudios GWAS pueden combinarse para aumentar el poder estadístico, y así poder identificar nuevos loci de riesgo. Recientemente, en un metaanálisis que incluye 2 de los GWAS previos y un tercero más reciente realizado con una nueva cohorte de 1.831 casos y 2.546 controles y replicado en 4.064 casos y 4.685 controles de Michigan, Toronto, Terranova y Alemania53, se han identificado 3 nuevos loci de susceptibilidad, NOS2, FBXL19 y próximo a PSMA6-NFKBIA. Todos ellos se asocian tanto con psoriasis cutánea como con artritis psoriásica. Asimismo, en este mismo estudio se ha replicado la asociación con una señal próxima a RNF114 que había sido descrita recientemente49,51.
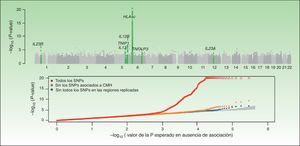
Concepto integrador del componente genético y la inmunopatogenia de la psoriasisComo ilustran los resultados del estudio CASP (fig. 1), el MHC constituye el principal determinante de la susceptibilidad genética en la psoriasis. Se han descrito varios SNP en la región del MHC que se asocian con la psoriasis en diferentes GWAS. El SNP que presenta una asociación más intensa, rs1219187, presenta un marcado desequilibrio de ligamiento con el alelo HLA-Cw*060228, el principal alelo de riesgo54, pero existen otras señales independientes tales como rs202254426, rs2073048, localizado en c6orf10, un potencial mediador de la vía del TNF-α y rs13437088, a 30kb55 de HLA-B en dirección al centrómero y a 16kb de MICA (MHC class i polypeptide-related sequence A precursor) en dirección al telómero54. Un análisis detallado en 2 poblaciones chinas de etnia han ha identificado una asociación de HLA-B*57 con un aumento del riesgo de psoriasis, y de HLA-B*40 con una disminución del mismo, de forma independiente de HLA-Cw*0602 y el locus C6orf1054. La segunda asociación se ha validado recientemente en un estudio que demuestra que MICA*016 aumenta el riesgo de desarrollar psoriasis sin artritis, y la homozigosidad para MICA*00801 aumenta el riesgo de desarrollar artritis en pacientes con psoriasis56.
En esta figura se resumen los resultados del GWAS realizado por el CASP. En la figura superior se representan los valores de significación estadística en relación a su posición cromosómica. Este tipo de gráfico se conoce como gráfico «Manhattan», puesto que las regiones de alta significación estadística se asemejan a la vista de una ciudad con rascacielos. En este caso, los estudios de replicación confirmaron la asociación de 7 regiones marcadas en el gráfico de color verde. En el gráfico inferior, conocido como «QQplot», se ordenan los valores de significación (esto es, observed P-value) y se comparan con la distribución teórica en ausencia de asociación (es decir, expected P-value). Este gráfico permite inspeccionar rápidamente la existencia de SNP asociadas a la enfermedad ya que, en ausencia de asociación, los valores deberían situarse sobre la diagonal. En este caso se puede observar cómo el QQplot que incluye las SNP de la región HLA (rojo) se desvía claramente. Al excluir esta región (naranja) y las otras regiones asociadas (azul) se puede ver cómo el gráfico se aproxima al valor esperado (zona sombreada). En ambos gráficos la significación para la región HLA-C se ha truncado para facilitar la interpretación de los resultados.
Fuente: Elder et al.26; Nair et al28. GWAS: Genomewide Association Studies; CASP: Collaborative Association Study of Psoriasis.
La psoriasis guttata, que se asocia con HLACw6 en hasta el 100% de los casos57, frecuentemente viene precedida por una faringoamigdalitis estreptocócica (raramente por dermatitis perianal, balanoprostitis o vulvovaginitis), pero infrecuentemente por otras infecciones estreptocócicas de la piel, como impétigo o erisipela. En el transcurso de una faringoamigdalitis se produciría una presentación de antígenos estreptocócicos o superantígenos mediada por HLA-Cw6 a los linfocitos T naïve de las amígdalas, que proliferarían, se diferenciarían a fenotipo efector y de memoria y adquirirían capacidad de direccionamiento (homing) cutáneo (CLA+)26,54, mientras que el peptidoglicano de la pared estreptocócica podría activar alternativamente a los linfocitos mediante la activación de los receptores Toll-like mediada por citocinas58. Con el tiempo se produciría una expansión oligoclonal de linfocitos T dirigidos contra antígenos estreptocócicos y con direccionamiento cutáneo, que empezarían a reconocer autoantígenos epidérmicos, dando lugar a lesiones de psoriasis en placas59. La predisposición genética determinada por HLA-Cw6 y otros componentes del MHC vendría dada por una pérdida de tolerancia frente a autoantígenos epidérmicos, especialmente si su expresión se encuentra aumentada o aparece (neoantígenos) en la psoriasis. Es el caso de diversas proteínas, como las queratinas K16 y K17, la β-defensina-4 (codificada por DEFB4), la psoriasina (S100A7), calgranulina (S100A8 y S100A9), las small proline-rich region proteins (SPRR) y las proteínas del LCE, muchas de las cuales se codifican en el complejo de diferenciación epidérmica localizado en 1q21.3 (PSORS4). Los genes que codifican las β-defensinas humanas (péptidos antimicrobianos con actividad semejante a citocinas) se localizan en varios clusters; uno de ellos, localizado en la región 8p23.1, se ha demostrado recientemente que se asocia con una CNV tipo amplificación de los genes DEFB4, DEFB103 y DEFB104, que codifican las β-defensinas 4, 3 y 2, respectivamente60.
Como la inmensa mayoría de linfocitos T en el infiltrado de la psoriasis no presentan expansión clonal, deben intervenir otros mecanismos independientes de antígeno, en los que también puede participar HLA-C actuando como ligando de los killer immunoglobulin-like receptors (KIR). Estos receptores regulan la actividad de las células NK-T y son codificados por el gen KIR, cuyo locus se ha asociado con la psoriasis y la artritis psoriásica61–65.
Otro importante determinante genético de la psoriasis tendría que ver con la regulación de la vía de señales NF-kB (fig. 2). A20 y ABIN1, productos de los genes TNFAIP3 y TNIP1, respectivamente, intervienen en la destrucción mediada por ubicuitina del complejo IKKγ/NEMO y otros componentes de la vía de transducción de señales del TNF-α. Recientemente se han desarrollado modelos murinos con ablación específica de la expresión de A20 en la piel y los macrófagos que desarrollan una dermatitis psoriasiforme66 y un cuadro de artritis67. Estos modelos experimentales confirmarían la trascendencia de esta vía en la patogenia de la psoriasis y el papel de A20 como inhibidor de la activación de las células dendríticas y la respuesta autoinmune68,69.
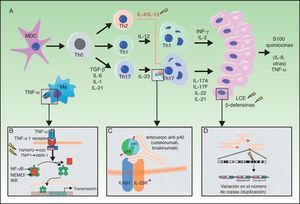
A. Principales vías patogénicas en la psoriasis con implicaciones genéticas: las células dendríticas mieloides, tras activarse por diversas citocinas tales como TNF-α, IFN-α, IFN-γ e IL-6, producen IL-12 e IL-23. Los linfocitos T naïve, en presencia de factor de crecimiento tumoral (TGF) β, IL-6, IL-1 e IL-21, se diferencian a linfocitos Th17, que expresan el receptor de IL-23 y proliferan en presencia de esta citocina. Los linfocitos Th17 producen IL-17A, IL-17F, IL-22 e IL-21, que activan a los queratinocitos desde el punto de vista inmunológico y proliferativo, dando lugar a la producción de TNF-α, IL-1, IL-6, IL-8, S100A7 y otras proteínas S100 y péptidos antimicrobianos (β-defensinas). A). La unión de TNF-α a su receptor activa una cascada de señales que da lugar a la liberación de NFκB de su complejo inhibitorio NFκB essential modulator/inhibitor of κB kinase (NEMO/IKK), lo que determina la transcripción de A20, un regulador negativo de la NFκB potenciado por la proteína ABIN-1. La psoriasis se asocia con determinados polimorfismos en los genes que codifican estas 2 proteínas inhibitorias. B). La inhibición de la señalización mediada por IL-23 constituye el mecanismo de actuación de los anticuerpos monoclonales inhibidores de la p40, como ustekinumab. La psoriasis también se asocia con polimorfismos de los genes que codifican las subunidades p19 y p40 de la IL-23 y la IL-12/IL-23, respectivamente, así como una subunidad del receptor de IL-23. C).Se ha descrito la asociación de CNV en los genes de proteínas del LCE y β-defensinas humanas con la psoriasis. CNV: copy number variants; LCE: late cornified envelope; MDC: las células dendríticas mieloides.
Fuente: modificada de Duffin et al.45.
Las alteraciones en la transcripción de las citocinas de la familia Th2 (IL-13, IL-4, IL-5) interferirían con la regulación negativa de la diferenciación de los linfocitos T naïve a Th1770, así como de la síntesis de IL-17 por los mismos71. Curiosamente, aunque se han identificado señales relacionadas con el gen IL-13 y el cluster que regula la transcripción de diversas citocinas Th2 en diversos estudios48, recientemente se ha restringido dicha asociación a los pacientes con artritis psoriásica43.
Últimos avancesComo prueba del carácter dinámico del estudio de la genética de la psoriasis, y a modo de conclusión, baste citar los dos últimos avances en este campo, que presentan relación con formas monogénicas de psoriasis pustulosa.
La psoriasis pustulosa generalizada se caracteriza por la aparición de episodios repetidos de erupción pustulosa generalizada acompañada de fiebre alta, leucocitosis y niveles elevados de proteína C reactiva, que puede asociarse con psoriasis en placas, y de la que se ha descrito una variante hereditaria de transmisión autosómica recesiva. Estudiando 9 familias tunecinas con esta enfermedad se ha identificado un ligamiento con un intervalo de 1,2 megabases en 2q13-q14.1 y una mutación homozigota en el gen IL36RH, que codifica el antagonista del receptor de la IL-36 (una citocina con propiedades antiinflamatorias), y determina la síntesis defectuosa de una variante menos estable y potente en cuanto a su interacción con el receptor IL1 receptor-like 2. Como consecuencia de esta mutación tiene lugar un aumento en la producción de IL-8 y otras citocinas proinflamatorias por parte de los queratinocitos, que determinan el cúmulo intraepidérmico de polimorfonucleares72. Otras mutaciones del mismo gen se han identificado en tres casos esporádicos de la misma enfermedad73, que ha pasado a conocerse como deficiency of the IL-36R antagonist (DITRA) y presenta grandes analogías con la deficiencia del antagonista del receptor de IL-1 (DIRA), una enfermedad autoinflamatoria descrita en 2009 que responde de forma drástica al tratamiento con antagonistas de la IL-1, como anakinra74.
El otro avance significativo ha sido la identificación del gen CARD14 como responsable de la asociación con PSORS2, al identificarse mutaciones determinantes de un aumento de función de la proteína transcrita (caspase recruitment domain-containing protein 14) en 2 familias extensas, una europea, con múltiples casos de psoriasis y artritis psoriásica en un 30% de ellos, y otra de Taiwan, así como una mutación de novo en una niña con psoriasis pustulosa esporádica de inicio precoz75. En otro artículo, el mismo grupo de autores describen 15 variantes adicionales de CARD14, su distribución en 7 cohortes de pacientes con psoriasis (más de 6.000 casos y controles), sugestiva de epistasis con HLA-Cw6, y su efecto sobre la activación de NF-κB y la transcripción de diversos genes en queratinocitos transfectados con los diversos mutantes76. La caspase recruitment domain-containing protein 14 se localiza habitualmente en las capas basal y suprabasal de la epidermis, mientras que su expresión en las lesiones de psoriasis pasa a estar aumentada de forma difusa y localizada en las capas suprabasales. Las mutaciones de CARD14 asociadas con el desarrollo de psoriasis determinan un aumento de la activación de NF-κB y la expresión de diversos genes asociados a la psoriasis en los queratinocitos.
En ambos casos estos hallazgos refuerzan las hipótesis patogénicas actualmente vigentes sobre el papel de los queratinocitos en la patogenia de la psoriasis, y amplían nuestro conocimiento sobre los mecanismos de producción de lesiones pustulosas, así como de las excepcionales formas monogénicas de la enfermedad.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEste manuscrito se ha realizado en el contexto del Proyecto Estratégico PSE-010000-2006-6 con financiación del Ministerio de Ciencia e Innovación.
Conflicto de interesesLos autores declaran que no tienen ningún conflicto de intereses.