El proyecto europeo BIOMED-2 Concerted Action BMH4-CT98-3936 describe protocolos estandarizados de amplificación por reacción en cadena de la polimerasa (PCR) de los diferentes loci de los genes del receptor de la célula T (TCR) e inmunoglobulinas (Ig) con el objetivo de obtener una mayor sensibilidad y especificidad en el estudio de clonalidad de las neoplasias linfoides. En el estudio de clonalidad T, además del TCRγ se propone el estudio adicional de los reordenamientos del gen TCRβ y del locus TCRδ (útil en casos de neoplasias de células Tγδ+). En el estudio de clonalidad B, junto al segmento FRIII del gen IgH se diseña el estudio de otros segmentos (FRI, FRII), así como de los genes Igλ e Igκ o reordenamientos incompletos DJ del gen IgH y κde (kappa deleting element). La lectura de los resultados de la amplificación se realiza mediante sistemas automatizados de lectura (GeneScan) o mediante el sistema heterodúplex.
The European Biomedicine and Health (Biomed-2) Concerted Action Project BMH4-CT98-3936 has defined standardized protocols for polymerase chain reaction (PCR) amplification of different loci of the T-cell receptor (TCR) and immunoglobulin (Ig) genes with a view to achieving greater sensitivity and specificity in the assessment of clonality of lymphoid neoplasms. To assess T-cell clonality, analysis of TCRβ gene and TCRδ rearrangements (useful in cases of Tγδ + cell neoplasms) is proposed alongside that of TCRγ. For analysis of B-cell clonality, along with the framework (FR) III segment of the IgH gene, other segments are studied (FRI, FRII) in addition to Igλ and Igκ genes or incomplete DJ rearrangements of the IgH gene and the κ deleting element. The results of the amplification are read using automatic reading systems (GeneScan) or using a heteroduplex system.
En los últimos años, en los laboratorios de Biología Molecular y servicios de Patología se han incorporado a la rutina diagnóstica una serie de técnicas de biología molecular
basadas en la técnica de la reacción en cadena de la polimerasa (polymerase chain reaction [PCR]) con el objetivo de demostrar la presencia de una proliferación linfoide monoclonal T o B dominante en infiltrados cutáneos linfoides atípicos (análisis genotípico)1-3. Los resultados del análisis genotípico, interpretados en un contexto clínico-patológico apropiado, constituyen una herramienta importante en el diagnóstico de los procesos linfoides malignos (linfomas), especialmente en aquellos casos en los que los hallazgos morfológicos e inmunofenotípicos no son concluyentes4,5.
Las técnicas de PCR pueden realizarse no sólo a partir de muestras en fresco o congeladas, sino también a partir de biopsias de archivo incluidas en parafina, precisando únicamente la conservación de fragmentos de ADN relativamente pequeños (> 100–200 pares de bases [pb])1. Esto supone una ventaja incuestionable respecto a otras técnicas, como la técnica de Southern Blot que precisa de fragmentos de ADN de gran tamaño (> 1.000 pb), lo que limita su estudio a material en fresco o congelado6,7. Los métodos basados en técnicas de PCR evitan la exposición a material radiactivo, permiten una mayor rapidez en la obtención de resultados y menor tiempo de trabajo de laboratorio. Las diferencias metodológicas existentes entre los distintos laboratorios difieren fundamentalmente en la selección de los oligonucleótidos cebadores y en los sistemas de lectura o análisis de los fragmentos de PCR amplificados2,8-14.
El proyecto europeo BIOMED-2 Concerted Action BMH4-CT98-3936 se ha diseñado con la finalidad de desarrollar unos protocolos estandarizados de PCR para el estudio de clonalidad linfoide B y T en el diagnóstico de los procesos linfoproliferativos. Define diferentes protocolos para múltiples cebadores dirigidos frente a distintos loci de los genes del receptor de las células T y B en un intento de alcanzar una mayor sensibilidad y especificidad en la detección de clonalidad linfoide T y B, respectivamente15,16.
Reordenamiento de los genes TCR/IgHEl receptor de la célula B es una inmunoglobulina (Ig) específica para cada célula formada por 2 cadenas pesadas y 2 cadenas ligeras (kappa [k] o lambda [λ]). El receptor de la célula T (TCR) está formado por la unión o bien de una subunidad alfa (α) con una beta (β) (fenotipo α/β) o bien una subunidad gamma (γ) con una delta (δs) (fenotipo γ/δ). Los genes que codifican las Ig y el TCR contienen diferentes segmentos variables (V), de diversidad (D) y de union ( J) que sufren un proceso de reordenamiento durante las primeras fases de la diferenciación de la célula linfoide17,18.
Este proceso de reordenamiento genético se inicia con la unión de uno de los posibles segmentos D a uno de los posibles segmentos J. Posteriormente se produce la unión de uno de los segmentos V en el caso de los genes IgH, TCRβ y TCRδ (fig. 1). En el caso de los genes Igλ, IgK, TCRα y TCRγ se produce la unión directa entre los segmentos V y J (fig. 2). Este reordenamiento sigue un orden jerárquico. En la diferenciación hacia una célula B, primero se reordena el gen IgH, seguido del gen IgK, produciéndose la deleción de éste para formar una célula Igλ+. En la diferenciación hacia una célula T, primero se reordena el gen TCRδ, luego el TCRγ seguido por el TCRβ con posterior deleción del TCRδ y finalmente se reordena el gen TCRα. El reordenamiento funcional de los genes Ig y TCR genera la expresión de los distintos receptores (Ig, TCRγδ o TCRαβ) en la superficie celular. Estas configuraciones TCRγδ o TCRαβ aparecen en las células T postímicas, siendo la configuración TCRγδ la menor fracción18.
Reordenamiento VDJ del gen IgH. Las regiones V del gen IgH están formadas por tres regiones hipervariables o«complementary determining regions»[CDR] I, CDRII y CDRIII. Estos segmentos CDR se alternan con cuatro segmentos de poca variabilidad denominados framework (FR) sobre los que se hibridan los cebadores de la PCR (flechas).
El gen IgH (locus 14q32.3) dispone de 46–52 VH segmentos funcionales dependiendo del haplotipo de cada individuo, los cuales pueden agruparse en 6–7 subgrupos o familias según su homología. Además, existen 27 segmentos DH funcionales agrupables en 7 familias (DH1-7) y 6 segmentos funcionales JH. El gen IgK(locus 2p11.2) contiene múltiples VK agrupados en 7 familias y 5 segmentos funcionales JK. El gen Igλ (locus 22q11.2) dispone de 73–74 Vλ agrupables en 11 familias y 7 Jλ (4 funcionales Jλ1, Jλ 2, Jλ 3 y Jλ7) que codifican los 4 isotipos de Igλ17,19.
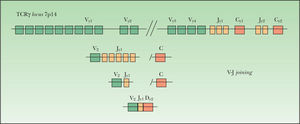
El locus TCRλ (cromosoma 7p14) tiene 14 segmentos genéticos variables Vγ (10 funcionales o reordenables), 5 segmentos de unión Jγ agrupados en 2 grupos Jγ1 (Jγp1, Jγp y Jγ1) y Jγ2 (Jγp2 y J72) y dos regiones constantes Cγ1 y Cγ2. Los segmentos Vγ se separan en cuatro subgrupos según su secuencia homóloga: el grupo I (Vγ1-8) contiene 5 segmentos funcionales y 4 pseudogenes; los grupos II, III, IV están formados por un solo segmento Vγ9, Vγ1O y Vγ11. El gen TCRβ (7q34) se forma por reordenamiento de uno de los 65 segmentos Vβ (39–47 de los cuales son funcionales y se agrupan por su homología en 23 familias) con uno de los segmentos Jβ (2 familias, Jβ1-2)18.
La existencia de estos múltiples segmentos V, D y J combinables hace que se produzca un repertorio de combinaciones posibles de 2×106 en el caso de las Ig, 3×106 posibles reordenamientos TCRαβ y 5×103 moléculas TCRγδ. Por otro lado, estas posibilidades combinatorias aumentan su diversidad mediante la inserción o deleción de nucleótidos en las áreas de unión de los segmentos, lo cual supone un total repertorio de moléculas de Ig y TCR superiores a 1012. Finalmente, en el caso de la célula B la variabilidad puede aumentar aún más mediante un proceso de hipermutaciones somáticas en el segmento V del gen IgH y de las cadenas ligeras de las células B maduras a su paso por el centro germinal de los folículos linfoides, lo cual aumenta asimismo la diversidad y especificidad antigénica17-19.
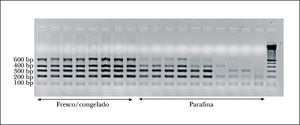
Aspectos técnicos del proyecto BIOMED-2Control de calidad del ADNEl control de calidad del ADN es un aspecto crucial en la aplicación del programa BIOMED-2, principalmente a muestras incluidas en parafina. Se aconseja el desarrollo de una PCR para amplificar genes control con 5 pares de cebadores que permiten amplificar fragmentos de 100, 200, 300, 400 y 600 pb de longitud, lo cual nos orientará sobre la calidad del ADN de la muestra analizada. Todos los fragmentos de PCR del proyecto BIOMED-2 miden menos de 600 pb (fig. 3). Se recomienda realizar la PCR con 2 cantidades de ADN para excluir el efecto de inhibidores de la PCR (tabla 1)15.
Protocolos de reacción en cadena de la polimerasa (PCR) BIOMED-2
| En el protocolo BIOMED-2, para la PCR-VH se emplean 3 sets de cebadores, cada uno con 6 o 7 cebadores, un set para cada FR (FRI, FRII, FRIII), capaces de unirse a los correspondientes VH (VH1-7). Se emplea un simple set JH capaz de unirse a los 6 JH funcionales. El producto PCR-Vh esperado es de aproximadamente 100–170 pb, 250–295 pb y 310–360 pb para FRIII, FRII y FRI, respectivamente. La pCr-Dh emplea 7 cebadores, uno por familia, y el mismo set JH de la PCR-VH. El producto de PCR esperado es de un tamaño entre 100–420 pb. La longitud del gen VJ de IgK varía entre 120–300 pb y para Igλ de 140–165 pb. Además, el locus Igk puede sufrir el reordenamiento del segmento Kde directamente al VK, de una longitud de 210–390 pb. Este reordenamiento produce la inactivación del alelo Igk. El tamaño de los productos de la PCR del TCRγ varía entre 231–283 pb para el grupo I, 147–207 pb para el grupo II, 132–182 pb para el grupo III y 116–165 pb para el grupo IV. El tamaño del producto final del reordenamiento β es de aproximadamente 170–325 pb, dependiendo del grupo de cebadores empleados |
| Reactivos de la PCR |
| 100ng ADN |
| 10 pmol de cada primer (marcados con fluorocromo o no dependiendo del sistema de lectura que se emplee). El número y tipo de primer o cebador para cada tubo depende de cada PCR específica |
| Nucleótidos para una concentración final de 200μM |
| Cloruro de magnesio (MgCl2) a una concentración final de 1,5mM |
| Taq polimerasa 1–2 unidades (dependiendo de si el tubo de PCR tiene más o menos cebadores) (AmpliTaq Gold [Applied Biosystems, Foster City, CA, USA]) |
| ABI Buffer II o ABI Gold para un volumen final de 50μl (AmpliTaq Gold [Applied Biosystems]) |
| Condiciones de la PCR |
| Preactivación de la Taq polimerasa durante 7 minutos a 95°C |
| Temperatura de hibridación 60°C |
| Número de ciclos: 35 |
| Fases por ciclo:Desnaturalización 30 (a 45) segundos (equipos nuevos de PCR son más rápidos)Hibridación 30 (a 45) segundosExtensión 30 segundos (a minuto y medio) |
| Extensión final de 10 minutos |
| Mantenimiento posible del producto a temperatura ambiente o 15°C |
El material fresco o congelado es el mejor tipo de muestra para obtener ADN de calidad, sin embargo, la mayor parte de las muestras estudiadas en la rutina diagnóstica suelen corresponder a material fijado en formol y parafinado. El ADN obtenido de estas muestras suele ser de mala calidad, sobre todo si la biopsia fue archivada durante más de 2 años. El proceso de fijación seguido y la calidad del tejido estudiado son asimismo factores determinantes20. Por ello, resulta fundamental realizar un control de calidad del ADN previo a la amplificación. Si el ADN obtenido no supera en el control de calidad una longitud en pb superior al tamaño del gen o segmento amplificado, los resultados obtenidos no suelen ser valorables. Se aconseja asimismo la práctica de un control histológico simultáneo a la obtención del tejido para la extracción del ADN para corroborar que la muestra estudiada es representativa.
Análisis de los fragmentos de reacción en cadena de la polimerasaRecientemente, se han descrito sistemas automatizados de lectura de los fragmentos de PCR amplificados que permiten aumentar la capacidad de discriminación con respecto a los sistemas clásicos de lectura en geles de electroforesis21-28. El proyecto BIOMED-2 recomienda la lectura de los productos amplificados de PCR mediante electroforesis capilar utilizando cebadores marcados con fluorocromos (GeneScan [GS]) o utilizando geles de poliacrilamida en los que se analiza la formación de heterodúplex (análisis heterodúplex [AH])29-31.
En una neoplasia linfoide, al proceder todas las células de un clon maligno éstas presentarán el ADN reordenado de la misma forma, por lo que al estudiar mediante PCR el reordenamiento VDJ se obtendrá uno o 2 productos de PCR (reordenamiento de uno o 2 alelos). Por el contrario, si el ADN procede de una población policlonal existirán numerosos reordenamientos VDJ. La visualización del producto de PCR se puede realizar mediante electroforesis en geles de acrilamida o electroforesis capilar. En el primer caso observaremos múltiples bandas discretas si se trata de una población policlonal o una o dos bandas si se trata de una población monoclonal. Mediante electroforesis capilar observaremos múltiples picos con una distribución gaussiana de pesos moleculares en el caso de poblaciones policlonales y uno o dos picos en las poblaciones monoclonales.
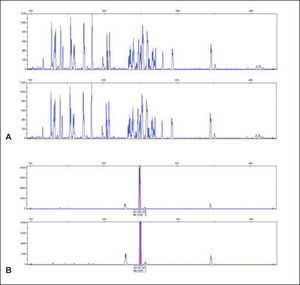
El AH utiliza productos de PCR de doble cadena y los separa en función de su longitud y conformación, mientras que GS analiza productos de una sola cadena y los separa únicamente en función de su longitud. En el caso de una muestra con una población linfoide monoclonal se formará una proporción variable de homodúplex. Su visualización mediante electroforesis en gel de poliacrilamida permite ver una banda de una longitud adecuada. Las muestras con una elevada proporción de células policlonales producen elevada cantidad de heterodúplex con poca probabilidad de unión de homodúplex.
El GS permite distinguir entre los productos amplificados incluso cuando difieren en un solo pb de longitud, permitiendo detectar un mismo clon en diferentes muestras o tejidos sin necesidad de realizar una secuenciación. Se considera que el sistema AH podría mostrar una sensibilidad y especificidad superiores al GS, especialmente en las amplificaciones de los reordenamientos de IgK, Kde e Igγ. En el análisis de los segmentos DJ del gen IgH pueden usarse indistintamente AH o GS15.
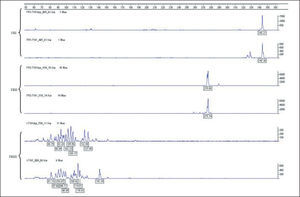
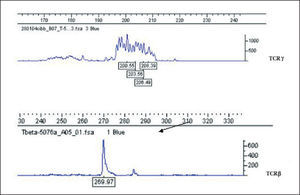
Análisis de fragmentos fluorescentes (GeneScan)En el sistema GS uno de los cebadores es marcado con fluorocromo y después de la amplificación los productos de PCR se discriminan o se separan en sistema de electroforesis en capilares de polímeros. Para dicho proceso, un microlitro del producto de PCR se mezcla con 0,5μl de un producto de peso molecular estándar (GeneScan 400-ROX, Applied Biosystems, Foster City, CA) en 9μl de formamida desionizada (Applied Biosystems) para su separación por desnaturalización en cadenas simples de ADN. Los productos son detectados de forma automática por lectura de fluorescencia mediante un sistema láser en un secuenciador automático de ADN (ABIPrism 3100, Applied Biosystems). Un resultado se considera monoclonal si se obtienen uno o 2 picos de producto amplificado con una longitud en pb apropiada para la técnica de PCR que se está analizando. Si se detectan de 3 a 5 picos puede considerarse como un resultado oligoclonal y más de 5 picos de longitudes diferentes como un resultado policlonal. Para considerar un resultado como un patrón monoclonal verdadero, la altura de un pico debe ser de al menos el doble de la distribución normal de los picos policlonales, correspondientes a las células reactivas acompañantes del infiltrado (fig. 4).
La sensibilidad global en la detección de clonalidad linfoide B o T de los protocolos BIOMED-2 y lectura mediante GS es de aproximadamente 0,5-5%. Los límites de la sensiblidad dependen de la técnica empleada, de la calidad de la muestra (las muestras parafinadas tienen menor rendimiento) y de la proporción de la población linfoide reactiva acompañante. Una técnica se considera suficientemente sensible si detecta al menos una población clonal que suponga el 5-10 % de la población linfoide total del infiltrado.
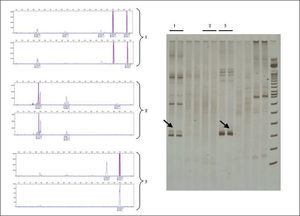
Análisis de heterodúplexLos productos de PCR se desnaturalizan por calor (95°C durante 5 minutos) y posteriormente se renaturalizan rápidamente a temperatura baja (4°C durante 1 hora) para buscar de nuevo su hibridación espontánea, ahora de forma aleatoria, de tal modo que se obtienen fragmentos de ADN homo- o heterodúplex con diferente conformación. Las diferentes conformaciones obtenidas dan distintas características físicas a los heterodúplex, lo que permite separarlos mediante electroforesis en geles de poliacrilamida no desnaturalizantes. Los homodúplex logran una hibridación perfecta y emigran de forma más rápida. Los productos de PCR correspondientes a muestras monoclonales forman una elevada proporción de homodúplex mezclados con heterodúplex obtenidos de los linfocitos policlonales o reactivos acompañantes y se separan claramente en el gel de poliacrilamida en forma de una banda. Las muestras policlonales producen únicamente heterodúplex (fig. 5).
La técnica de AH es rápida y sencilla y posee una sensibilidad global en la detección de clonalidad linfoide B o T (aplicando los protocolos BIOMED-2) de aproximadamente el 1-10 %, incluso cuando se estudian biopsias de archivo incluidas en parafina.
Sensibilidad y especificidadFalsos negativos (sensibilidad)Los fallos en la detección de clonalidad pueden explicarse cuando el peso molecular del fragmento que se quiere amplificar es mayor que el que indica la PCR del control de calidad del ADN. El empleo de múltiples cebadores dirigidos contra diferentes segmentos diseñados en el proyecto BIOMED-2 permite minimizar los resultados falsos negativos como consecuencia de errores en la hibridación por la falta de especificidad u homología de los cebadores, particularmente en el estudio de clonalidad B donde las hipermutaciones somáticas en el segmento VH son las responsables de este fallo de hibridación19.
Falsos positivos (especificidad)La detección de falsos positivos o pseudoclonalidad es particularmente un problema importante en casos en los que el infiltrado linfoide es de características reactivas, con escasa celularidad, o cuando se estudian muestras de pequeño tamaño32. Este es un elemento crucial en los protocolos de BIOMED-2 y para minimizar este fenómeno se aconseja realizar el análisis por duplicado y si es posible estudiar más de una muestra del mismo paciente. Algunos autores recomiendan que este tipo concreto de muestras deben analizarse simultáneamente por el sistema GS y AH, dado el mayor índice de falsos positivos de GS en tales situaciones.
En aquellos casos en los que no se repita el mismo pico de clonalidad deberían ser considerados como resultados negativos o pseudoclonales. También resulta recomendable comprobar por duplicado la naturaleza clonal de un proceso linfoproliferativo mediante la detección de un clon dominante sobre diferentes dianas o segmentos genéticos de las Ig o del TCR.
Finalmente, conviene recordar que la detección de un reordenamiento monoclonal del gen TCR o Ig no implica inequívocamente un marcador de un linaje o estirpe celular concreto. Si se excluye la posibilidad excepcional de un linfoma compuesto, se pueden detectar o bien reordenamientos aberrantes dobles T y B en una misma muestra o bien reordenamientos cruzados (infidelidades de linaje), sobre todo en neoplasias de células inmaduras, aunque también hasta en el 5-10 % de las neoplasias de células maduras33.
Estudio de clonalidad linfoide TAspectos técnicos y metodológicosLa amplificación del gen VJ de la subunidad TCRγ mediante PCR es el método más empleado en la evaluación de la clonalidad de un infiltrado cuando se sospecha un origen de estirpe celular T4,8,10-12,23,26,29,30,34,35. Es una técnica relativamente simple, ya que el gen se reordena empleando un repertorio limitado de segmentos genéticos. La longitud del segmento amplificado (aproximadamente de unos 200 pb) permite la obtención de resultados en la mayor parte de muestras de material de archivo. El gen TCRγ se encuentra reordenado en la mayor parte de las células T tanto si expresan en superficie el TCRαβ como el TCR78.15,36,37
El análisis de PCR del gen completo VDJ o incompleto DJ que codifica la subunidad TCRβ es una técnica de mayor complejidad9,38,39. La subunidad TCRβ posee un mayor número de segmentos potencialmente reordenables, por lo que obliga a utilizar un mayor número de cebadores, siendo los segmentos amplificables de mucha mayor longitud o tamaño (fig. 6). El gen TCRβ se encuentra reordenado no sólo en todas las células TCRαβ, sino también en muchas TCRγδ15,36,37. El análisis de PCR del gen TCRβ representa un estudio complementario a realizar cuando exista una sospecha razonable de linfoma de células T con resultados de PCR-TCRγ no concluyentes. La combinación de ambos estudios puede permitir la detección de clonalidad T en prácticamente el 100 % de las muestras de linfomas de células T15.
Pueden detectarse asimismo los reordenamientos del gen TCRδ y pueden ser de utilidad tanto en el estudio de neoplasias de células linfoides inmaduras como en los procesos linfoproliferativos derivados de células TCRγδ36,37. Como consecuencia de su complejidad (con un elevado número de segmentos posibles a combinar y muy separados entre sí en su distribución en el genoma), el gen TCRα no se incluye en el proyecto BIOMED-215.
Resultados en linfomas cutáneos de células TLa mayor parte de los trabajos publicados empleando sistemas de lectura de geles de electroforesis muestran tasas de clonalidad en linfomas cutáneos de células T (LCCT) en un rango variable del 40-90 % de las muestras35. Diferentes aspectos técnicos, tipo de sistema de lectura de los productos amplificados, características de las muestras (parafinadas o congeladas, proporción variable entre estadios precoces o avanzados) pueden explicar las diferencias en las distintas series. Suele detectarse con mayor frecuencia una proliferación linfoide T monoclonal en los casos de micosis fungoide (MF) en estadios avanzados y de síndrome de Sézary (SS) (90-100 %) en relación con lesiones de MF en fases precoces (50-70 %)8,2,23,26,29,30,34,35,38-42.
La aplicación de técnicas de PCR y sistemas de lectura de elevada sensibilidad y especificidad (GS, AH) permiten detectar monoclonalidad linfoide en torno al 60–70 % de las muestras de LCCT8,12,23,26,29,30,40-42. En la serie de
Ponti et al 162/194 (83,4 %) muestras de LCCT resultaron clonales mediante PCR para la amplificación del TCRγ y análisis mediante AH35. Resultados similares (84 %) fueron obtenidos por los mismos autores empleando el sistema GS34. Parece existir una buena correlación entre las características del infiltrado (densidad, epidermotropismo), inmunofenotipo (pérdida de antígenos de células T) y estadio con la detección de la presencia de un clon dominante. Excepcionalmente, Cherny et al41 sólo detectaron un 42 % de muestras clonales mediante AH en una serie de LCCT, sin embargo, este resultado parece poco representativo debido al reducido número de casos analizados (n=7).
La aplicación de los protocolos BIOMED-2 y análisis por GS ha demostrado tener una alta sensibilidad en el estudio de distintos procesos linfoproliferativos cutáneos de células T. Aunque faltan resultados sobre series amplias de muestras en parafina, se han detectado clones T en un 63–85 % de muestras cutáneas de MF/SS mediante PCRGS del TCRγ y en un 63–75 % mediante PCR-GS del TCRβ38,42.
La detección de un clon dominante en los procesos linfoproliferativos CD30+ es sensiblemente menor que en el resto de LCCT, quizás en parte debido a las características peculiares de un infiltrado linfomatoso que suelen tener una celularidad variada43-45. En nuestra experiencia, la mitad de las muestras de papulosis linfomatoide (PL) de material congelado suelen ser clonales, así como las dos terceras partes de los LCCT CD30+. En un trabajo reciente realizado sobre muestras parafinadas de PL y LCCT CD30+ mediante PCR-GS sólo se detectó un clon T en un 22 % de los casos (4/18) de PL, particularmente en aquellas formas con un porcentaje elevado de células atípicas. En dicho trabajo en las muestras de LCCT CD30+ la tasa de detección alcanza el 65 % de los casos46.
Estos resultados indican que la demostración de un clon dominante es una característica importante en el diagnóstico de un LCCT, siendo infrecuente la presencia de un patrón de reordenamiento policlonal. Sin embargo, también se ha detectado la presencia de un clon linfoide T en un número variable de casos de dermatosis inflamatorias crónicas consideradas benignas y con una relación incierta respecto del desarrollo de un verdadero LCCT39,47,48. Algunos autores han propuesto el concepto de«discrasias linfoides cutáneas»para designar a este grupo de procesos, que incluirían la pitiriasis liquenoide, algunos casos de dermatosis purpúricas pigmentadas, paniculitis lobulillares atípicas, ciertas reacciones a fármacos o incluso un grupo de entidades incluidas bajo el concepto de«parapsoriasis»(PP)49.
El diagnóstico diferencial entre las lesiones de PP, definidas como placas eritemato-descamativas persistentes sin criterios histopatológicos diagnósticos de MF, con respecto de las lesiones precoces de MF puede plantear importantes dificultades prácticas. Existe una cierta controversia con respecto a la tasa de detección de clonalidad T en estos casos. Algunos autores detectan clonalidad linfoide T en casi un 50 % de las muestras, proponiendo que estos casos deben considerarse bien como lesiones pre-MF, bien como verdaderas MF incipientes. Sin embargo, otros autores no detectan clonalidad en prácticamente ningún caso50-54. Probablemente la razón de estas discrepancias se deba a la falta de unos criterios homogéneos empleados en la definición del concepto de PP (tabla 2)55. En nuestra experiencia, con la aplicación de PCR para el TCRγ la tasa de detección de clonalidad en lesiones de PP es muy baja, menor del 5 %, mientras que aproximadamente el 70 % de los casos de MF en estadios precoces son clonales56.
Criterios para el diagnóstico de la micosis fungoide incipiente (International Society of Cutaneous Lymphoma)
| Criterios | Mayores (2 puntos) | Menores (1 punto) |
| Clínicos | 2 años | 1 año |
| Persistentes y/o progresivasMáculas o placas MASZona no expuestaVariada forma/tamañoPoiquilodermia | ||
| Histológicos | Ambos | Cualquiera |
| Infiltrado linfoide superficial +Epidermotropismo sin espongiosisAtipia | ||
| Moleculares | Presente | |
| Reordenamiento monoclonal del TCR | ||
| Inmunohistoquímicos | 1 año | |
| CD2, CD3, CD5<50% CD7<10%Discordancia epidermis/dermis |
El diagnóstico definitivo de micosis fungoide se realiza con una puntuación de 4 puntos o superior.
Finalmente, el análisis mediante GS permite la evaluación de la concordancia clonal entre muestras de distintos procesos linfoides que acontecen de forma simultánea o sucesiva en un mismo paciente (por ejemplo, lesiones de MF y de PL). En el caso de que se trate de un mismo clon, el pico monoclonal debe ser idéntico, de la misma longitud, pudiéndose evitar la realización de una secuenciación de los productos de PCR57,58.
Los datos publicados en la literatura referentes a la detección de un clon dominante en procesos linfoproliferativos reactivos T es muy variable (0–24 %)59-61, tratándose en general de trabajos que emplean sistemas clásicos de electroforesis. Ponti et al demostraron la existencia de un clon dominante hasta en un 2 % de muestras de una serie extensa de lesiones reactivas T, indicando una buena especificidad del AH (97,7%), siendo particularmente útil en la distinción entre eritrodermias de origen inflamatorio y SS35. La especificidad de los protocolos BIOMED-2 en el análisis de muestras cutáneas de origen o carácter reactivo es elevada42. En este sentido, ante la necesidad de confirmar la presencia de un clon T en un infiltrado linfoide reactivo, el estudio del gen TCRβ, por su mayor repertorio genético, quizá ofrezca una mayor especificidad39.
Estudio de clonalidad linfoide BAspectos técnicos y metodológicosEl estudio del reordenamiento del gen IgH se realiza mediante cebadores consensuados para las regiones FRI, FRII o FRIII del segmento VH y para el segmento JH. El estudio del segmento FRIII-JH es el más habitualmente realizado por razones de índole técnica, ya que el número de genes combinables es menor y ambos segmentos están muy próximos en el gen reordenado (aproximadamente 100 pb).
Dentro de un contexto clínico-patológico adecuado, en casos en los que no se detecta clonalidad linfoide B tras la amplificación por PCR del segmento FRIII-JH, se recomienda el estudio de los segmentos FRII y el FRI. Sin embargo, el segmento FRI tiene una longitud considerable y su estudio requiere muchos más cebadores, por lo que pocas veces es factible realizarlo a partir de muestras incluidas en parafina15.
En los protocolos BIOMED-2 se incluye no sólo el análisis del gen completo VDJ del gen IgH, sino también el diseño de una PCR para el estudio del reordenamiento incompleto DJ15. Este segmento se ve afectado con menor frecuencia por el proceso de hipermutaciones somáticas, fenómeno muy habitual en las neoplasias maduras de células B19. El elevado número de hipermutaciones somáticas, que ocurren en los segmentos VH durante el tránsito de la célula B por el centro germinal, condiciona una mayor probabilidad de fallo en la hibridación del primer y, por tanto, de resultados falsamente negativos.
Por otro lado, se describen las PCR para la amplificación de los genes Igλ e IgK que en determinadas circunstancias pueden permitir aumentar la sensibilidad en la detección de clonalidad B62. Además, no sólo puede realizarse el estudio del gen completo IgK, sino también la detección del reordenamiento del segmento Kde, tampoco afectado por hipermutaciones somáticas. El reordenamiento del Kde suele estar presente en todas las neoplasias Igλ y una tercera parte de las de origen IgK. El estudio de los reordenamientos incompletos DJ, así como de los genes Igλ e IgK y del Kde, se emplea en aquellos casos en los que no se ha podido demostrar la presencia de una población monoclonal B después del análisis del gen completo (VDJ) IgH (fig. 7)15.
Resultados en linfomas cutáneos de células BEl estudio de la clonalidad linfoide B mediante PCR de la región FRIII permite detectar clonalidad en un 30–70 % de los linfomas B dependiendo del tipo de muestra y técnica de laboratorio empleada1,27,63,64. La aplicación del protocolo BIOMED-2 en muestras de linfomas sistémicos de células B de calidad adecuada estudiando simultáneamente las regiones FRI, FRII y FRIII podría detectar un patrón clonal hasta en un 90–95 % de los casos. La razón por la cual no se detecta clonalidad en la totalidad de casos es, muy probablemente, la presencia de hipermutaciones somáticas15,16.
La combinación del estudio del reordenamiento completo de la IgH (VDJ) con el de los reordenamientos incompletos DJ, así como el reordenamiento de las cadenas ligeras, ha permitido alcanzar un porcentaje de detección de clonalidad en prácticamente el 100 % de los casos de los linfomas del manto, leucemias linfáticas crónicas, linfomas foliculares y los linfomas de la zona marginal, y hasta el 98 % de los linfomas difusos de células B grandes15,16.
En los linfomas cutáneos de células B (LCCB) los diferentes trabajos publicados que emplean técnicas de PCR sobre clonalidad B, y en general con sistemas de lectura en electroforesis, detectan una sensibilidad entre el 34–83 %42,63,65-70. Se ha de tener en cuenta que en la mayor parte de los trabajos publicados se analiza exclusivamente la región o segmento FRIII, por lo que los resultados falsos negativos pueden representar hasta el 50 %64.
Gellrich et al, en un trabajo reciente sobre muestras en parafina de LCCB y empleando los protocolos BIOMED-2 para la amplificación del gen completo VDJ IgH (FRIII, FRII, FRI), demostraron una tasa de clonalidad global del 55,8 % (el 100 % en los LCCB tipo de las piernas, el 54,5 % en los LCCB de la zona marginal y el 66,7 % en los LCCB foliculares)71. En este trabajo la PCR para FRIII detectó un 46,5 % de clonalidad (el 100 % en los tipo de las piernas, el 36,4 % en los linfomas de la zona marginal y el 40,7 % de los linfomas cutáneos foliculares). La PCR de FRI detectó sólo un 20,9 % considerando todas las muestras (hasta un 76 % de las muestras en parafina tenían un control de calidad inferior al aconsejable para amplificar el fragmento FRI). Sin embargo, si se consideraban sólo las muestras amplificables, la PCR de FRI mostraba la tasa de detección de clonalidad más alta (90 %), lo cual indica que es una excelente técnica, particularmente en material fresco o congelado. El estudio de otras regiones FR parece aumentar de forma significativa la sensibilidad y, probablemente aún más de forma muy considerable si se añaden asimismo en el análisis genotípico B los reordenamientos incompletos DJ, Igλ, IgK y Kde15.
ConclusiónEl proyecto de estudio de clonalidad BIOMED-2 y análisis mediante GS/AH ha demostrado ser una estrategia diagnóstica de elevada sensibilidad y especificidad en el estudio de pacientes con procesos linfoproliferativos cutáneos. El control de la calidad y amplificabilidad del ADN, particularmente cuando es obtenido a partir de material incluido en parafina, es un elemento crucial a la hora de establecer si una muestra en particular puede explorarse mediante una determinada técnica de PCR. El estudio de PCR debe realizarse por duplicado para cada muestra y, si es posible, en dos muestras diferentes del mismo paciente. Si no se reproduce el mismo pico de clonalidad (con igual longitud) no puede considerarse como un resultado positivo. De esta forma se evita la interpretación errónea de monoclonalidad (falsos positivos o pseudoclonalidad).
En el análisis de clonalidad de un infiltrado linfoide de células T, inicialmente se estudia la amplificación por PCR del TCRγ, ya que representa el prototipo de segmento de longitud corta y pocas regiones variables, lo que permite su preservación en material parafinado de archivo. El TCRγ está reordenado precozmente en la mayor parte de las neoplasias de células T, tanto en las de fenotipo TCRαβ como en las de TCRγδ. Debido a su mayor complejidad y laboriosidad técnica, el estudio del reordenamiento del gen TCRβ genera una estrategia complementaria para aquellos casos en los que ha fallado la detección mediante la amplificación del TCRγ.
La secuencia estratégica en el estudio de clonalidad B sería iniciar el estudio del gen IgH primero en su segmento FRIII y posteriormente los segmentos FRII y FRI. Aunque las frecuencias de la detección de reordenamientos incompletos DJ, IgK, Igλ, Kde son menores que en el estudio del gen completo VDJ, son dianas complementarias interesantes porque no se ven afectadas por las hipermutaciones, presentes particularmente en los LCCB de la zona marginal y los LCCB de células del centro folicular.
Conflicto de intereses
Declaramos no tener ningún conflicto de intereses.
Este trabajo ha sido financiado parcialmente por la beca FIS 03/394 del Ministerio de Sanidad y Consumo.











![Reordenamiento VDJ del gen IgH. Las regiones V del gen IgH están formadas por tres regiones hipervariables o«complementary determining regions»[CDR] I, CDRII y CDRIII. Estos segmentos CDR se alternan con cuatro segmentos de poca variabilidad denominados framework (FR) sobre los que se hibridan los cebadores de la PCR (flechas).](https://static.elsevier.es/multimedia/00017310/0000009900000008/v1_201304241514/S0001731008747574/v1_201304241514/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w9/t1/zx4Q/XH5Tma1a/6fSs=)